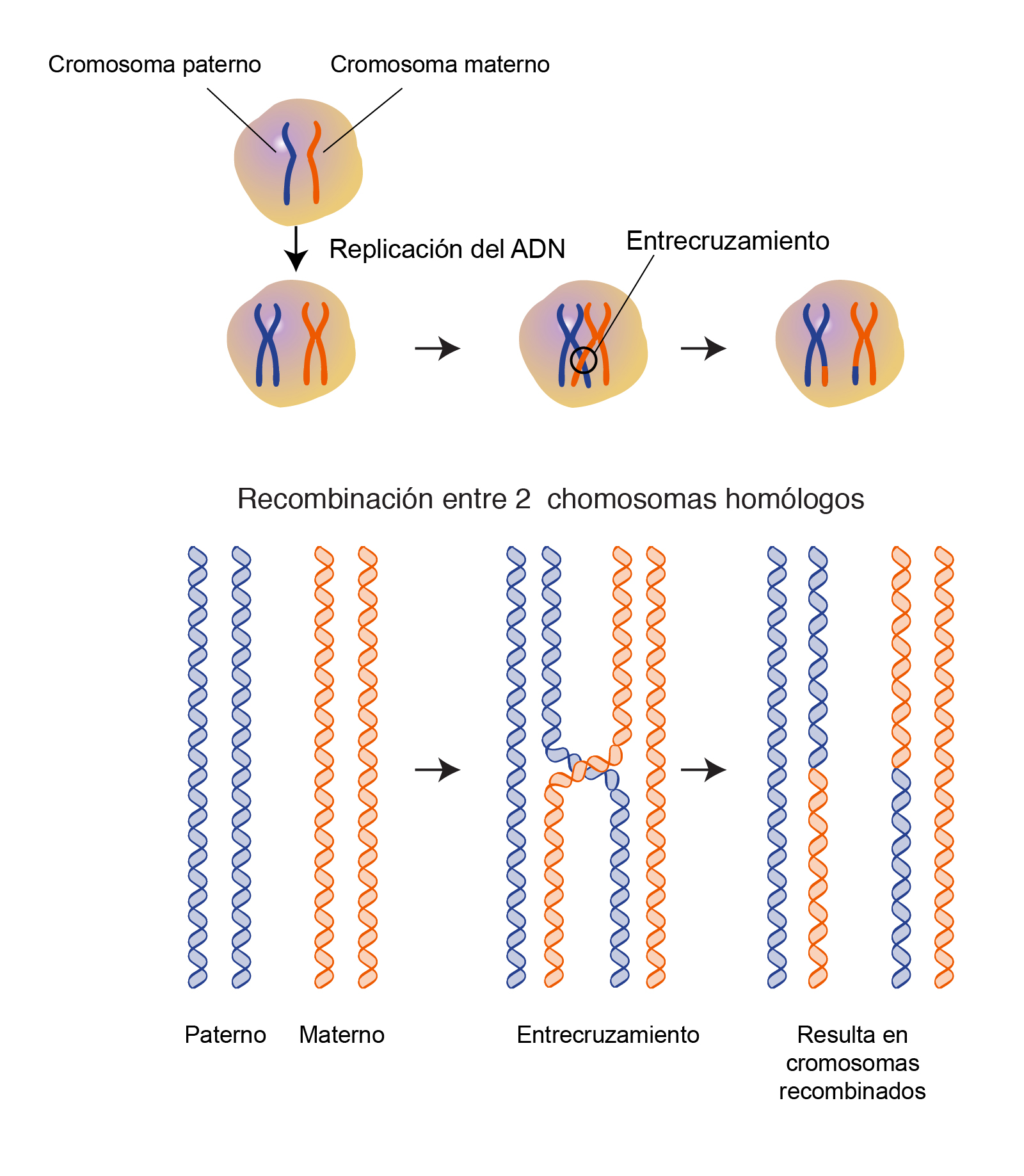
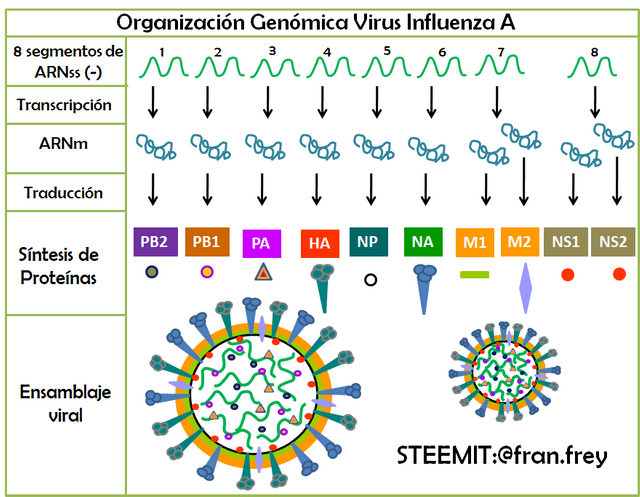
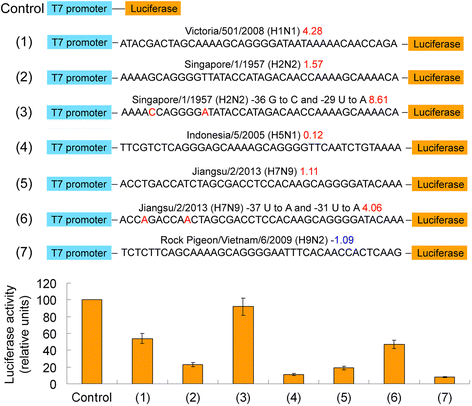
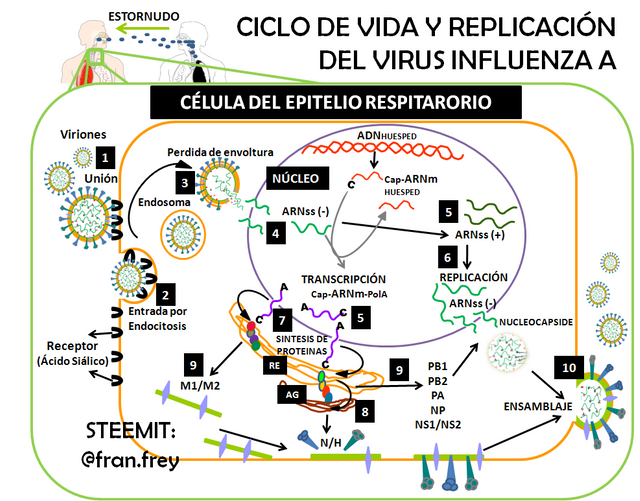
Las células madre de la vía aérea distal producen alvéolos in vitro y durante la regeneración pulmonar después de la infección por influenza H1N1
 |
| Imagen tomada con fines académicos de https://www.sciencedirect.com/science/article/pii/S0092867411011731 |
Clonación y seguimiento regional específico de células madre de Pulmón humano.
 |
| Imagen tomada con fines académicos de https://www.sciencedirect.com/science/article/pii/S0092867411011731#bib7 |
Bibliografía:
1. Kumar PA., Hu Y., Yamamoto Y., Hoe NB., Seok Wei T., Mu D., et al. Distal Airway Stem Cells Yield Alveoli In Vitro and during Lung Regeneration following H1N1 Influenza Infection. Cell Press [Internet]. 2011 [Citado el 29 dic 19]; 147 (3): 525-538. Disponible en https://www.sciencedirect.com/science/article/pii/S0092867411011731#bib7